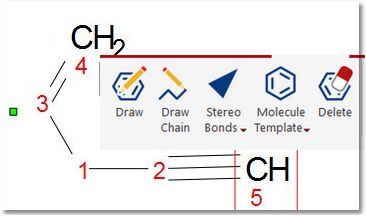
Mnova 包含一个基本的 "分子编辑器",该编辑器将在即将推出的软件版本中得到改进。该编辑器包括一个功能强大的工具栏,用于绘制分子:
通过 "绘制 "模式,您只需点击任何空白区域即可在波谱窗口中绘制碳原子。点击任何现有原子都会增加一个碳原子。如果点击单键,则会得到双键(点击双键则会绘制三键)。
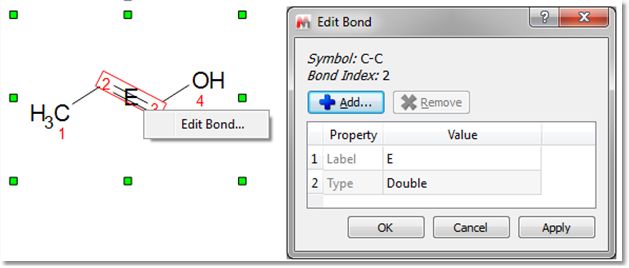
通过键的上下文菜单(右键单击键)也可以得到相同的结果。编辑键 "对话框允许您更改键的类型(单键、双键、三键、上键、下键)或设置键的标 签(例如添加立体化学符号):
您只需将鼠标悬停在原子上并输入相应的字母(如 "o "表示氧,"n "表示氮,"b "表示硼,"b "加 "r "表示溴,或更复杂的标签,如 Me、Ph、COOH 等)即可更改原子。请确保您在 "文件/首选项/分子 "中设置了键盘编辑选项。
您也可以通过双击原子显示 "编辑原子 "对话框来更改原子。在此您可以更改原子名称(例如从 C 改为 F)。
如果您的分子中同时加载了波谱,则需要在键入相应字母之前选择任何分子绘图工具(从 分子草图工具栏)(除非您激活了 "分子偏好设置",使 "键盘编辑 "功能始终可用)。更多信息请点击此处。
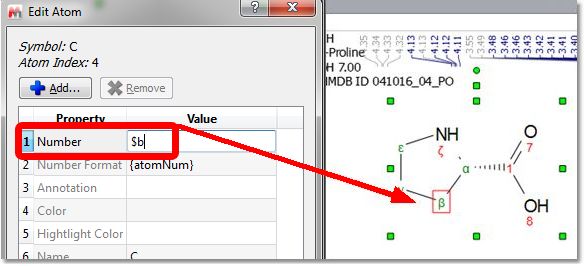
在 "编辑原子 "对话框中,您可以通过输入 $ 前缀为数字添加希腊字母;例如,$a, 4$b, 22$g$d21 将转换为 α, 4β, 22γδ21。
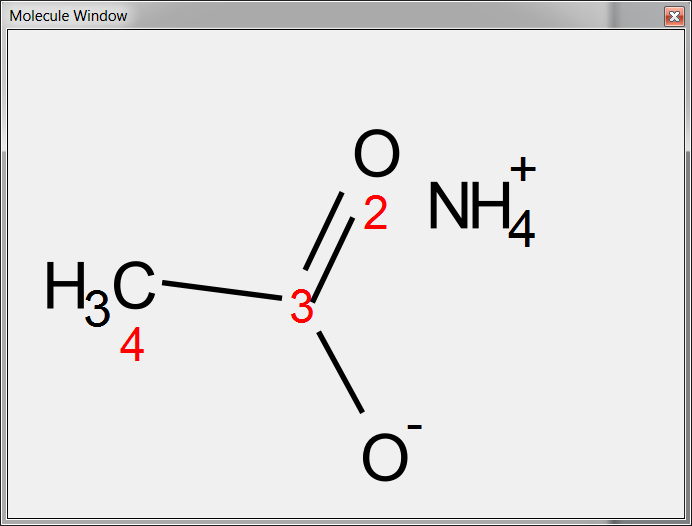
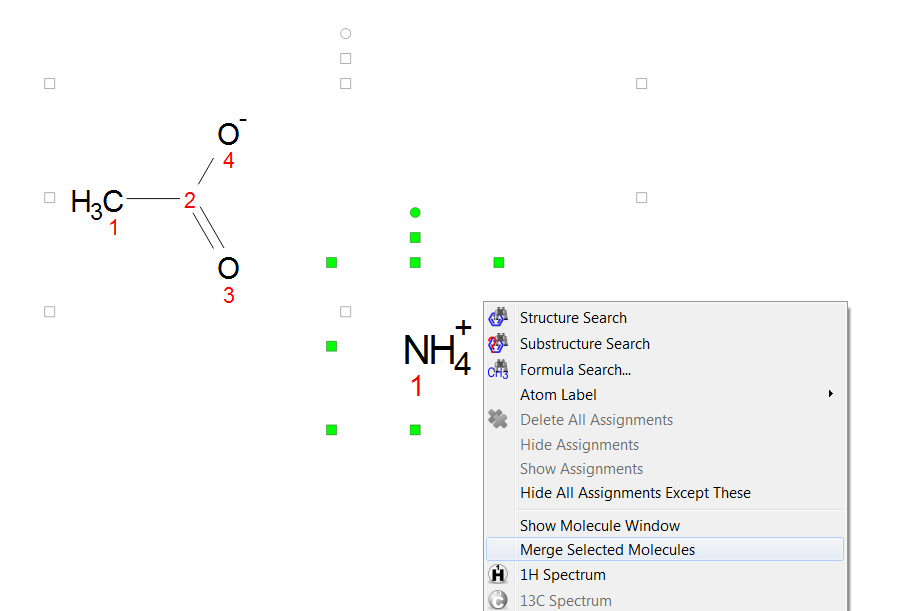
您可以在 "分子窗口 "面板("视图/面板/分子窗口")中绘制盐类,只需在其中绘制两个成分即可。要绘制 NH4+ 成分,您需要先绘制 CH4,然后将鼠标悬停在N后输入+(添加正电荷):
您可以通过绘制两个组分、选择它们、右键单击任意一个分子并选择 "合并所选分子 "来 得到相同的结果:
Mnova 不允许添加新原子(点击现有原子)或更改原子键类型(如果这会导致原子价超标), 但允许您通过拉伸现有原子键来创建新原子。在这种情况下,旧原子将被标记为无效化合价:
添加/删除新原子或更改化学键类型时,Mnova 也会自动更改原子价(例如氮原子的价为 3-5-3)。
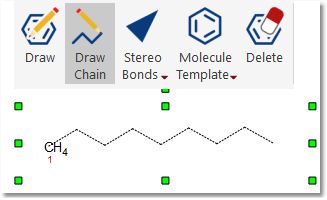
绘制链:点击此按钮后,通过单击、按住并拖动绘制链:
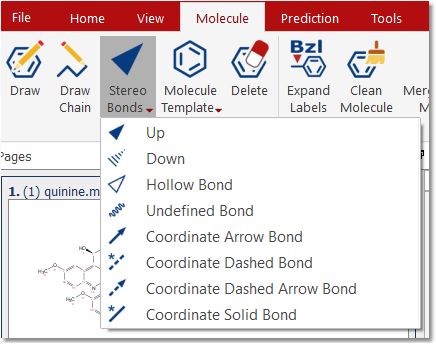
Stereo Bonds(立体键): 使用下拉菜单绘制立体键和坐标键 点击任何立体键都将改变其方向。
您可以在删除原子的同时删除原子键(按住 Ctrl/Cmd 键)。按住 Ctrl/Cmd 键的同时删除套索选中的多个原子,可以保留相邻的末端原子。
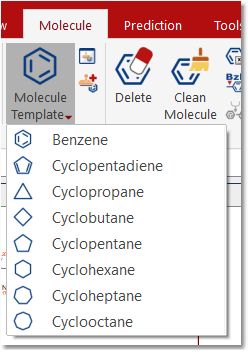
在 "分子模板 "下拉菜单中,您可以绘制循环:
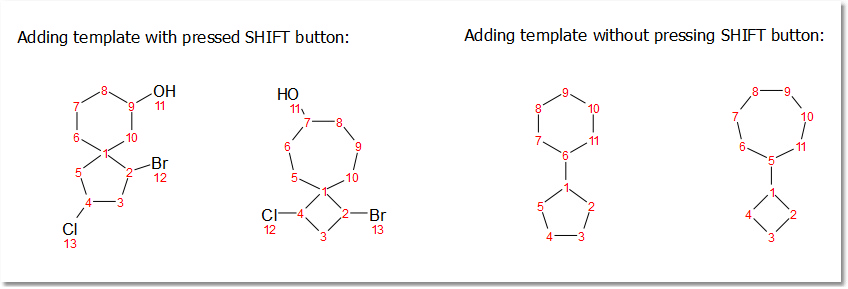
按住 "Shift 键 "的同时添加循环模板,即可绘制 Spiro 原子。
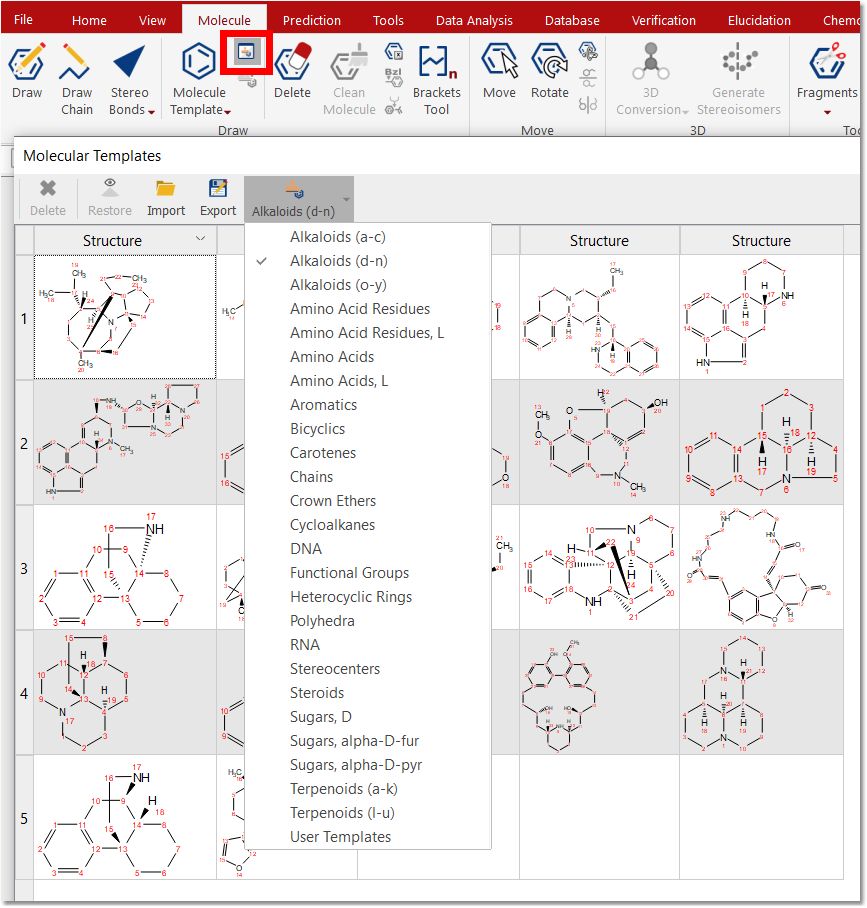
点击 "分子模板 "按钮可以加载更多模板。选择所需模板后,只需点击分子,然后再次点击波谱窗口即可粘贴分子结构。
您可以按住 shift 键并点击所需的原子,将任何模板添加为螺化合物。
您也可以通过点击 "Import(导入)"按钮并加载包含分子结构的 .SDF 文件或点击 "Molecule Ribbon(分子功能区)"的 "Add Molecule to user Templates(将分子添加到用户模板)"按钮来创建自己的用户模板。
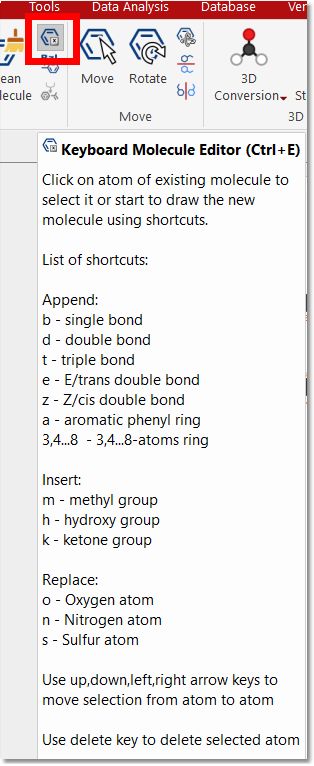
键盘分子编辑器 (快捷键:Ctrl+E): ,无需使用鼠标即可绘制分子。将鼠标悬停在按钮上可在工具提示中找到快捷键:
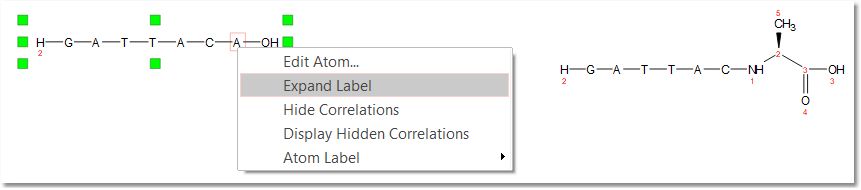
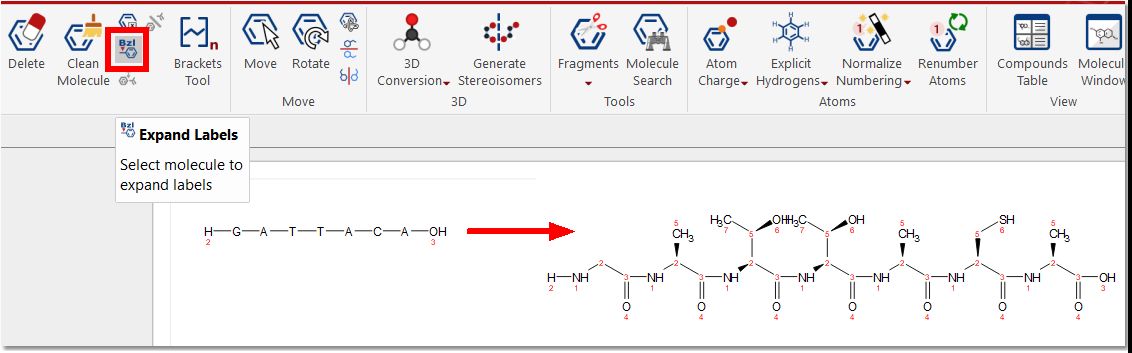
Expand Labels(扩展标签):用于绘制带有压缩基团的分子,并通过右键单击相应的基团来 扩展标签:
您可以通过启用 "Preferences/Molecule(偏好设置/分子)"中的 "Expand labels on importing(导入时展开标签)"选项以及点击分子工具栏中的 "Expand Labels(展开标签)"按钮来展开标签。
使用宏编辑器(在常规选项卡下)添加自由文本标签和分子属性中的几个参数。
分子标签存储在布局模板中,在应用模板时也会应用。另请参阅 "属性"章节(有关宏编辑器的更多信息)
合并所选分子(Merge Selected Molecules): 用于将多个片段合并为一个独特的结构。
Unmerge Molecules(解除 合并分子): 将合并的分子拆分成独立的结构。
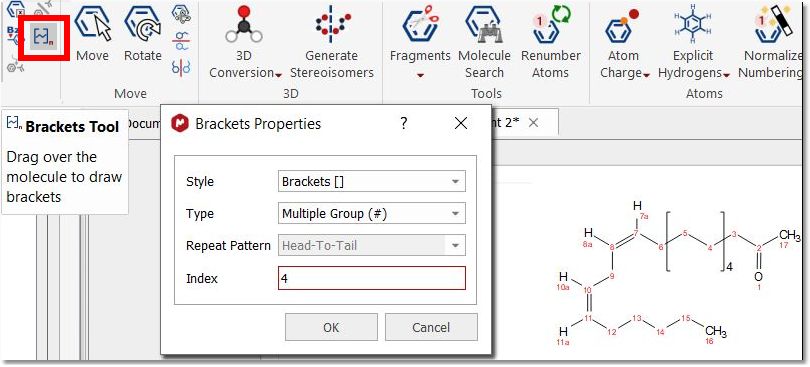
括号工具: 用于绘制聚合物。点击该按钮将显示一个包含不同括号选项(样式、类型、重复模式和索引)的对话框。
只有在打开该对话框时才能绘制括号。我们必须选择所需的属性,然后在分子的所需区域绘制括号。这样就可以在关闭对话框之前绘制任意数量的括号(必要时更改设置)。
在分子中绘制多个括号时,最后绘制的括号将以红色高亮显示,直到关闭 "括号属性" 对话框或绘制下一对括号为止:
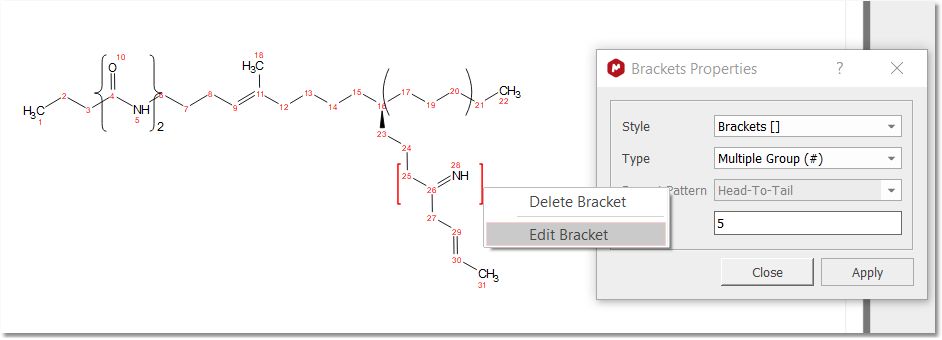
右键单击选定的括号,从右键菜单中选择 "Edit Bracket(编辑括号)",即可编辑该括号。该括号将以红色高亮显示,并显示 "括号属性 "对话框:
移动模式:该模式允许您通过点击和拖拽移动原子或原子键。
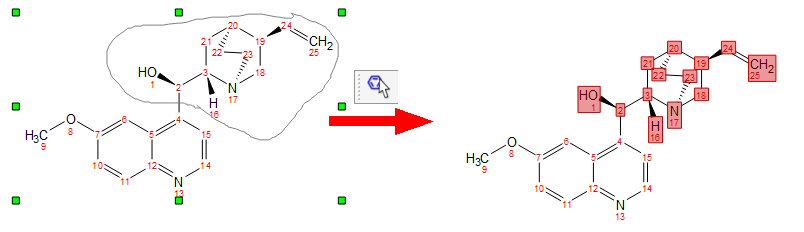
在该模式下,您还可以使用套索模式(在分子的所需部分点击并拖动鼠标)选择多个原子 (例如删除它们):
当您选择一个原子,将其移动到另一个原子上并松开鼠标键时,只有最后一个原子会保留 (除非第一个选择的原子为非碳原子,而第二个选择的原子为碳原子)。
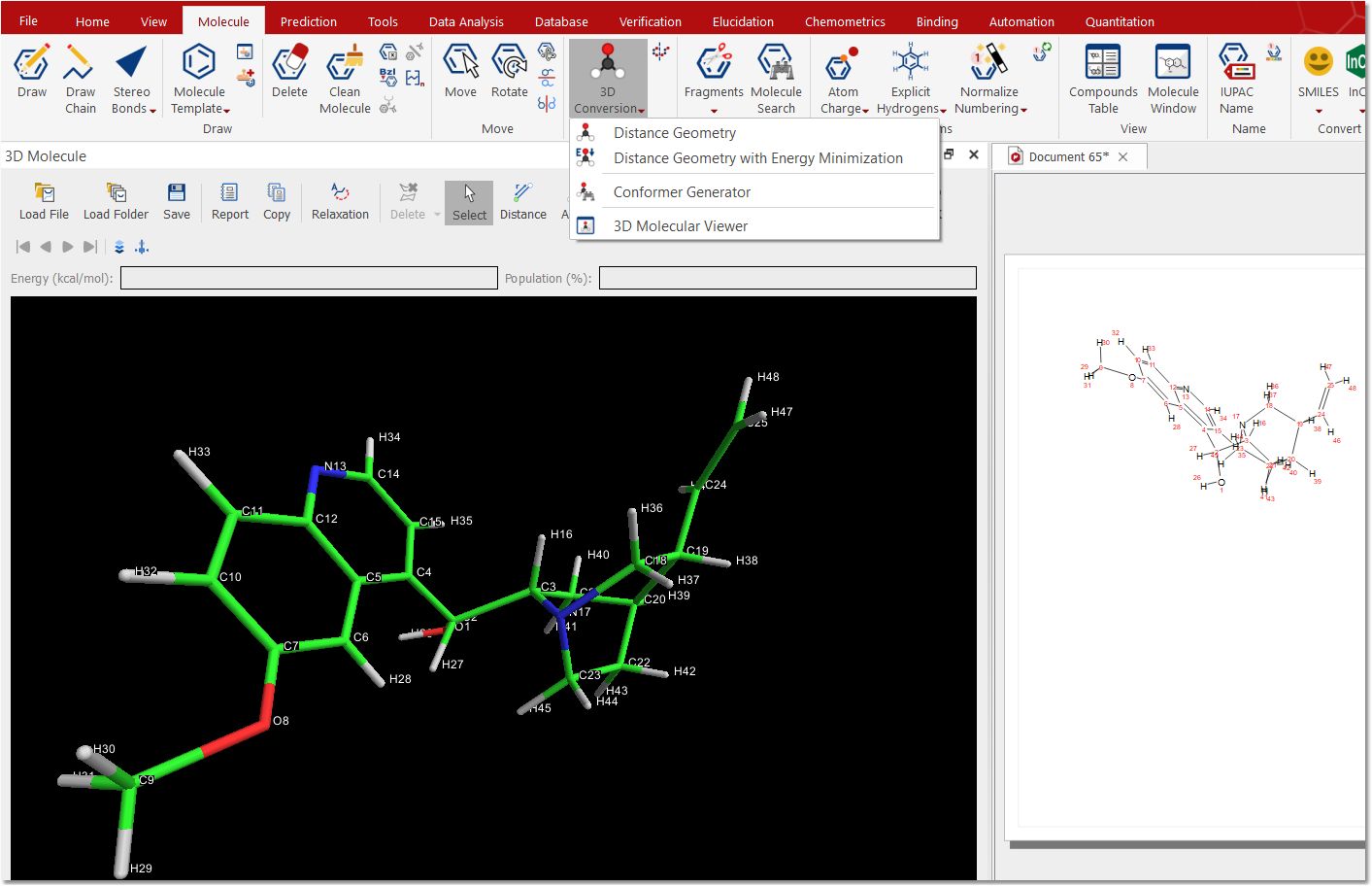
三维转换 该功能可将二维分子转换为三维结构,并计算每个构象的能量及其数量。
注意:如果无法在查看器中看到三维分子,请在 "首选项/界面"对话框中更改 OpenGL 引擎。
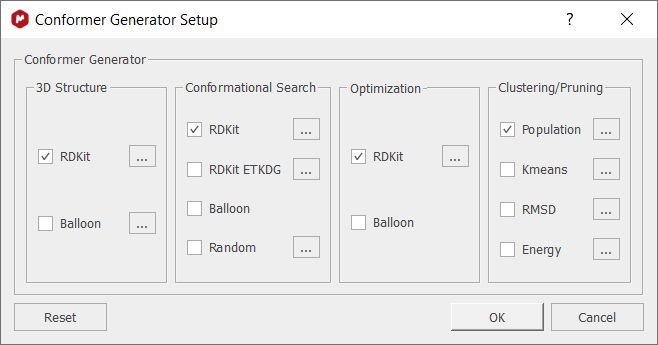
单击 "构象生成器 "选项可选择计算算法:
这样,用户就可以将不同的引擎(如 gmmx、rdkit 和 balloon)结合起来,创建 3D 构象列表。然后,可以通过均值聚类或 RMSD 缩减这些列表,从而生成最相关的构象的小列表。
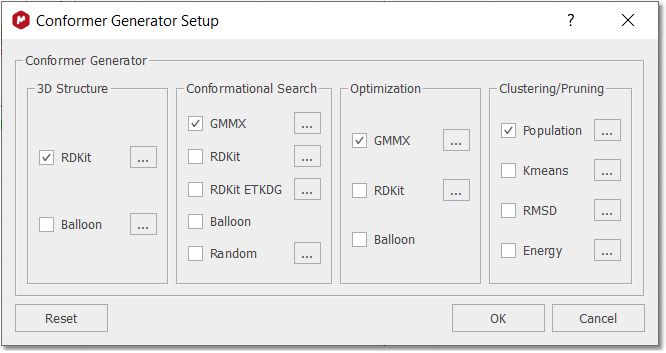
拥有Stereofitter许可证的用户可以使用 GMMX 构象搜索和优化功能:
对话框分为四个步骤:结构、构象、优化和聚类/运行。每个步骤都可以配置为使用不同的引擎运行,每个引擎也可以配置不同的选项。单击 "重置"(Reset)按钮后,将恢复默认的引擎组合以及每个引擎的默认选项。
加载 .mol 文件并使用任何可用选项时,所有可能的构象都将显示在 3D 分子面板中,包括每个构象的能量(kcal/mol)和数量(%)。
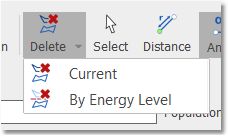
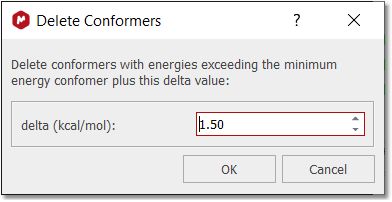
您只需点击 "删除当前构象 "按钮即可删除不需要的构象:
您还可以按能级删除构象:
3D 构象搜索 GMMX "工具需要获得 Stereofitter 插件的有效许可。
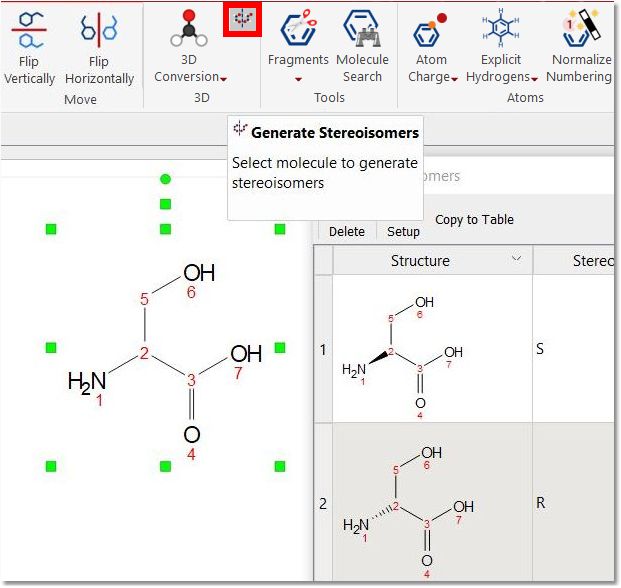
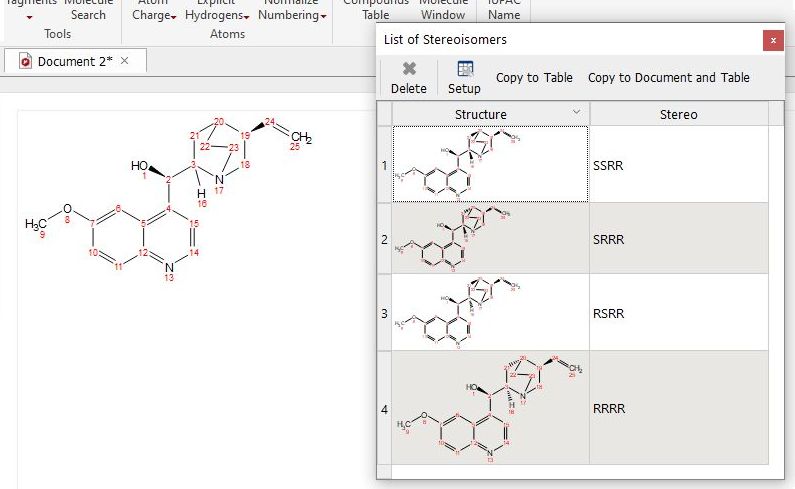
生成立体异构体: 它将显示给定分子结构(以普通键绘制)的所有可能立体异构体。
如果给定的分子包含任何立体键,则在计算立体异构体时将该键视为固定键。
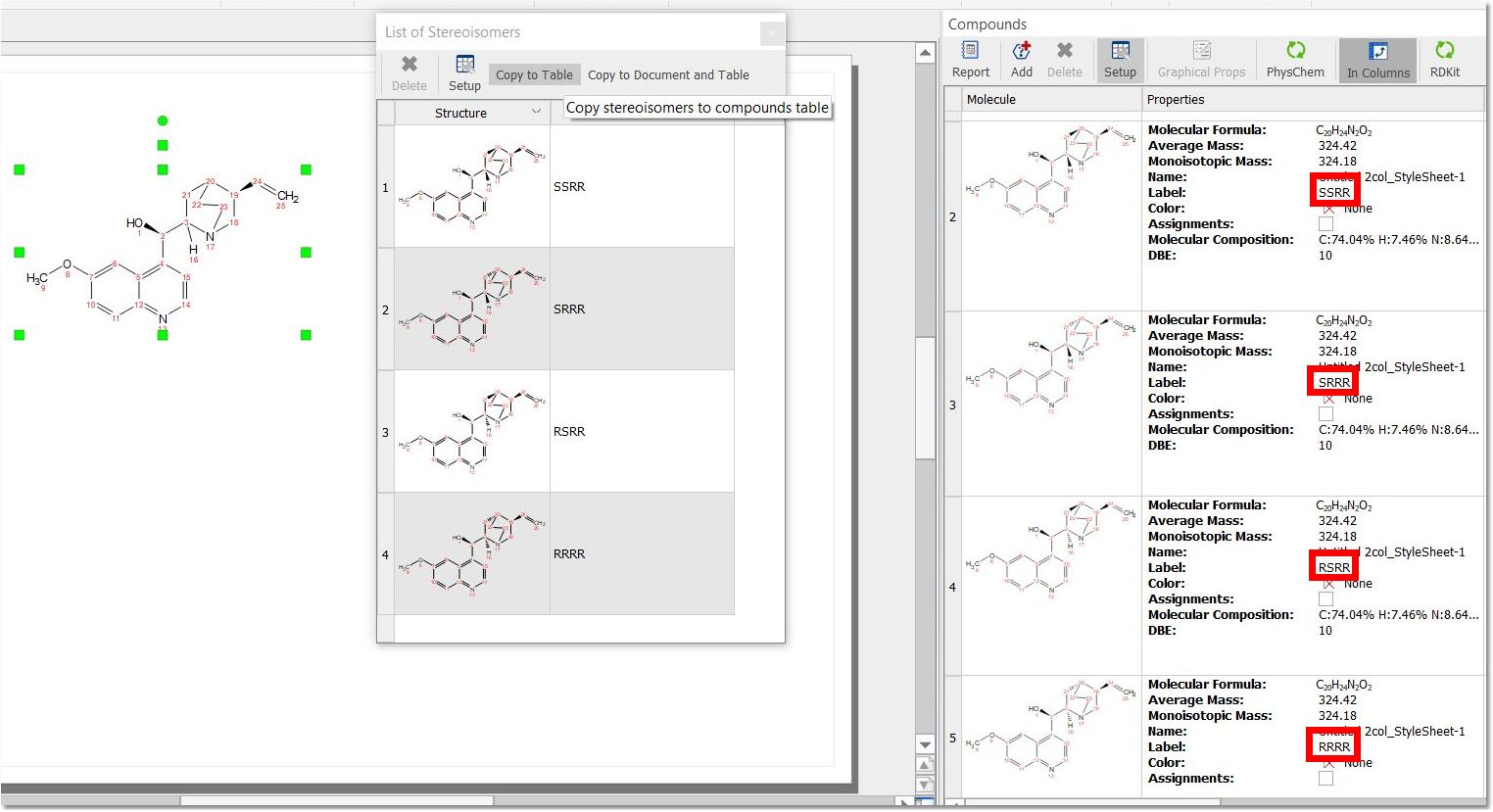
生成立体异构体后,将其复制到化合物表时,每个结构的立体信息都将添加到 "Label(标签)"标签中:
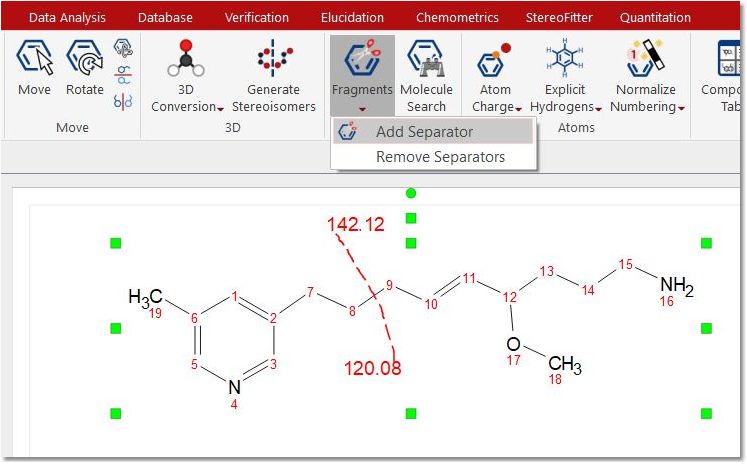
工具 该功能区允许您添加/删除分隔符,并在外部数据库(eMolecules 和 BMRB)中搜索分子。
选择 "Add Separator(添加分隔符)"后,鼠标将变为剪刀图标,您可以(按住左键)在 结构中画一条细线将其分隔。随后,分子上会显示一条红色虚线,并显示分割线上片段的分子质量。
您可以随意绘制分割线;选择 "移除分隔线 "工具后,所有的分割线将被一次性移除。
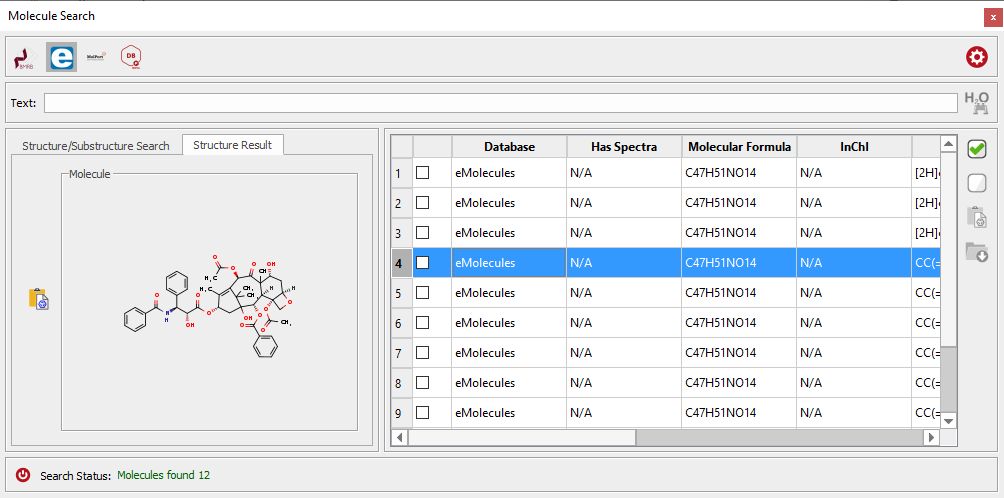
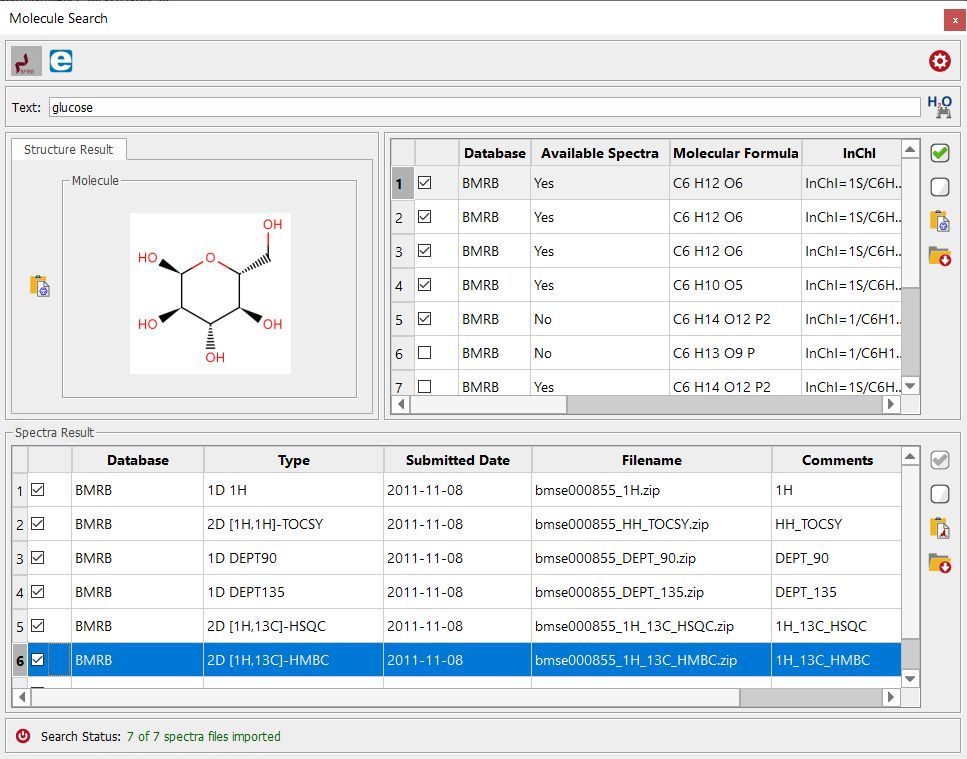
分子搜索 "功能允许您通过分子结构(或化合物名称)在 eMolecules 和 BMRB 数据库中进行搜索:
如果在数据库中找到命中的分子,您可以下载分子和波谱(如果有的话),并在 Mnova 中进行处理。
下面是 BMRB 数据库中葡萄糖数据集的示例:
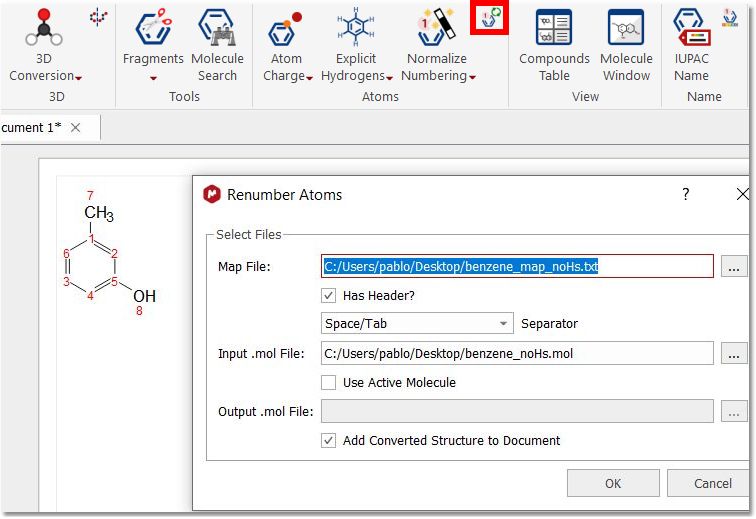
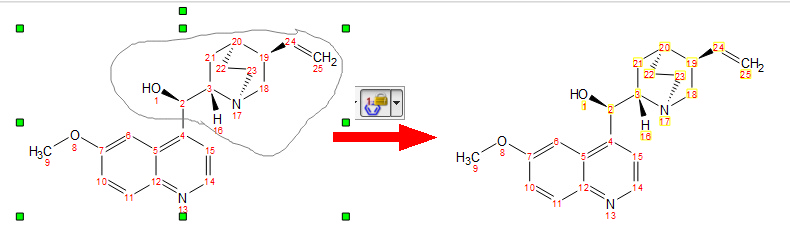
原子重新编号: 它需要一个地图文件、一个输入 .mol 和一个输出 .mol。输出将是原始的 .mol 文件,但所有原子编号都会按照映射文件中的定义进行更改。
映射文件应包含两列,第一列为当前编号,第二列为所需编号。下面是映射文件的示例:
输入 输出 1 1 2 2 3 5 4 4 5 3 6 6 7 7 8 8
请注意,您并不需要所有原子的完整列表,如果您想交换两个原子,可以使用这样的映射文件:
输入 输出 3 5 5 3
原子 该功能区包括添加正/负电荷(只需按 + 或 - 键即可为任何原子设置电荷)、自由基、添加/删除显式 H 以及归一化、锁定或连接编号等功能。
在基态模式下,第一次点击原子后,会出现单原子符号(- 符号)。第 2 次点击后,将显示二叉(Singlet)符号(-- 符号)。点击第 3 次后,将显示二重(三重)(^^ 符号)。最后,点击第 4 次后,根号将消失。
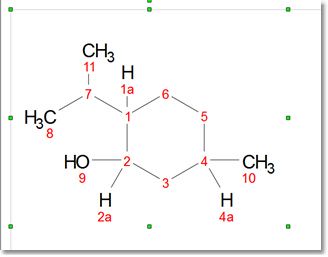
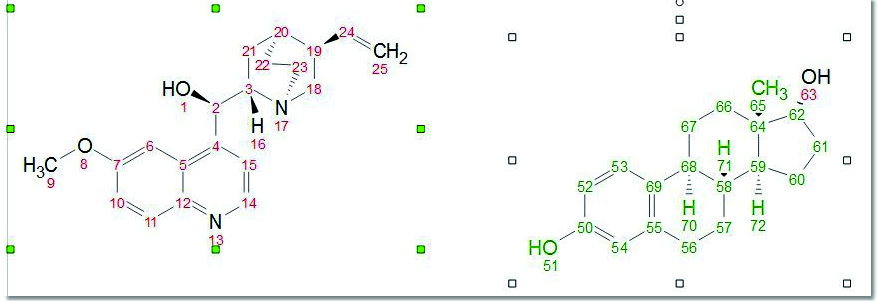
Normalize Numbering(归一化编号 )按钮对整个选定分子进行重新编号。 您可以 在 " 偏好设置 "中 选择是否要对导入(或粘贴)的 mol 文件(以及在连接片段时)自动重新编号;如果您在不同的应用程序中绘制了两个相等的分子,或以不同的顺序绘制,而您又希望对编号进行归一化处理,那么该功能将非常有用。
Normalize Explicit Hydrogen Numbering(显式氢编号规范化)"工具仅对显式氢进行编号,使其与重原子序数一致(附加 a、b、c...字母):
锁定编号 工具将固定所选结构部分的编号(使用套索)。被固定的原子将显示黄色背景:
点击 "Concatenate Numbering(合并编号)"按钮将删除文档中所有分子的重复原子编号。高亮显示的分子将被重新编号,这样原子编号就不会重复(从一个分子到另一 个分子之间会留有空隙)。分子重新编号的顺序取决于分子的大小(最大的分子将保留其编号)。
复制编号 "功能会将所选分子的编号作为模板,并将其应用到文档中与所选分子相同的所有分子。在处理具有不同编号的构象时,该功能对 Stereofitter 插件非常有用。
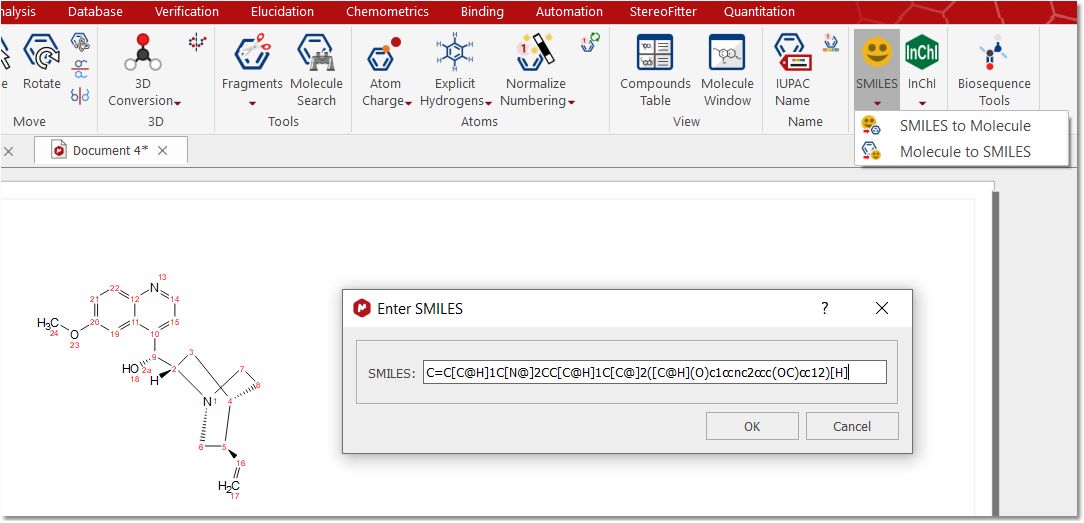
微笑 从剪贴板粘贴微笑字符串或点击 "Smiles to Molecule(将微笑转换为分子)"按钮后,Mnova 会将微笑字符串转换为适用的分子结构:
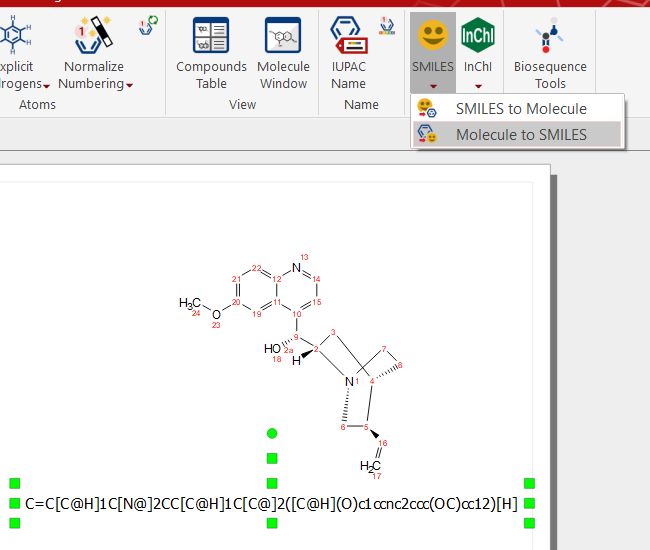
如果您有分子结构,则可以点击 "Molecule to SMILES(分子到 SMILES)"按钮获取微笑 字符串:
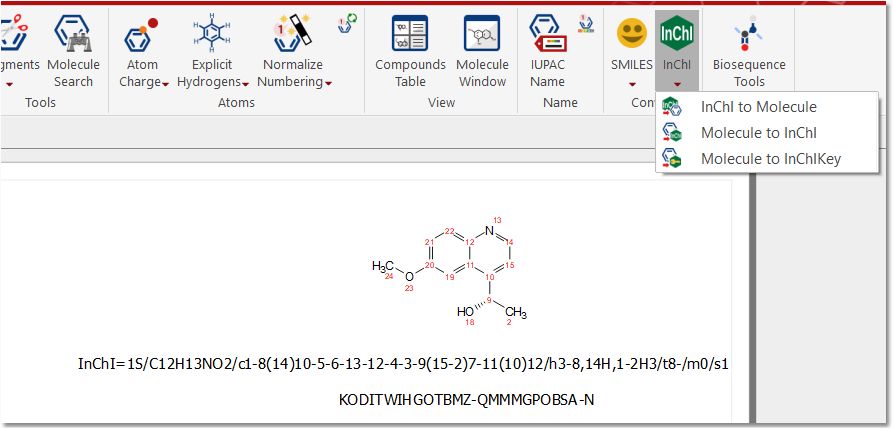
Inchi: 您只需将相应的 .inchi 文件载入 Mnova(或点击 "Inchi to molecule(Inchi 转分子)"按钮并粘贴 Inchi 字符串),即可将分子结构载入 Inchi 文件。
您还可以将分子结构转换为 Inchi 和 Inchi 键函数。
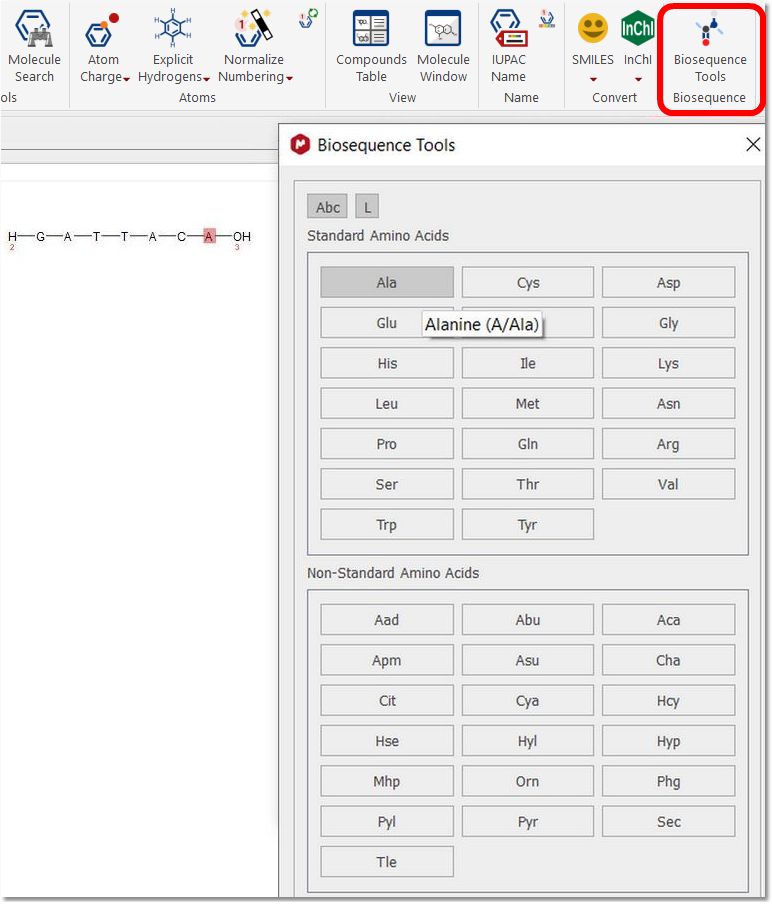
生物序列: 点击 "Biosequence Tools(生物序列工具)"按钮后,将显示一个对话框,允许您使用 20 种标准氨基酸和几种非标准氨基酸绘制肽:
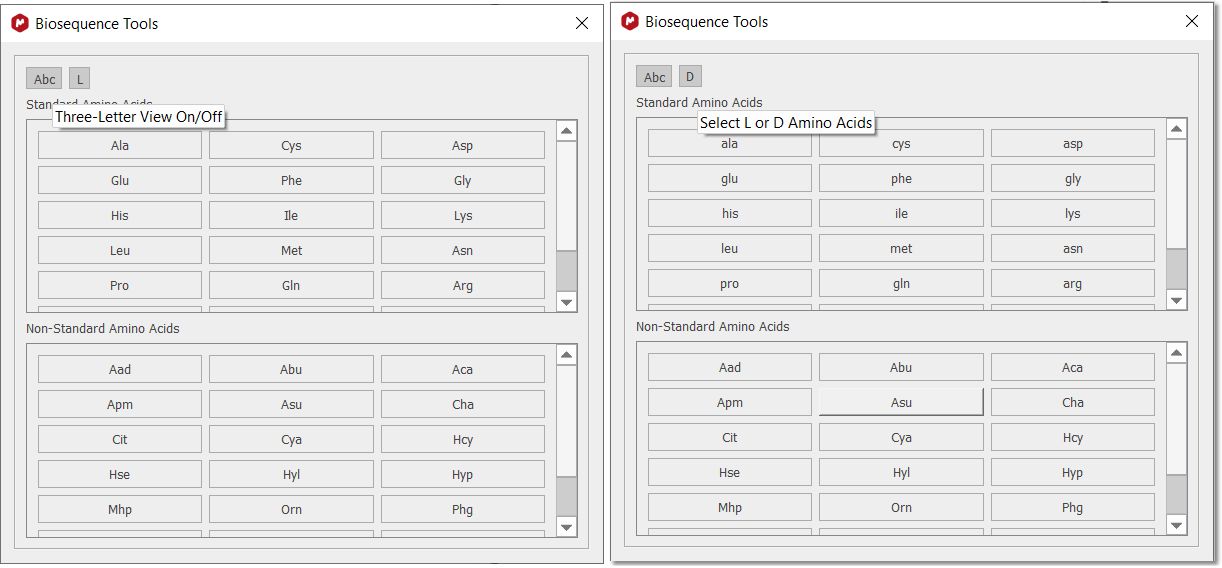
打开新的 "生物序列工具 "对话框后,光标将变为多肽模式,只能使用 1 或 3 个字母的代码绘制多肽。只需选中相应的切换按钮,就可以添加 D 或 L 氨基酸:
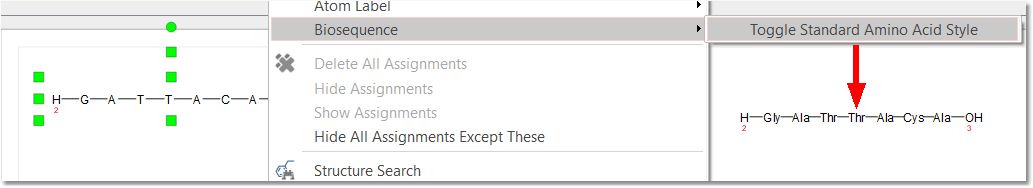
绘制肽链后,右键单击并从上下文菜单中选择相应选项,即可切换序列:
选择肽链中的 AA 并点击想要添加的 AA,即可在肽链中添加氨基酸。要删除氨基酸,只需从分子功能区选择 "Delete Atom/Bond(删除原子/键)"模式,然后点击氨基酸(将鼠标悬停在氨基酸上并按下删除键也可以)。
绘制肽链后,只需右击氨基酸并选择 "Expand Labels(扩展标签)",即可扩展氨基酸:
点击分子功能区的 "Expand Labels(扩展标签)"按钮,可以扩展整个肽序列:
|